MSdial
1 使用MSDIAL软件将质谱数据转换为Excel表格
1.1 创建project
点击左上角file,点击New project
1.2 Project file path
质谱数据在哪个文件夹,这里就选哪个文件夹。 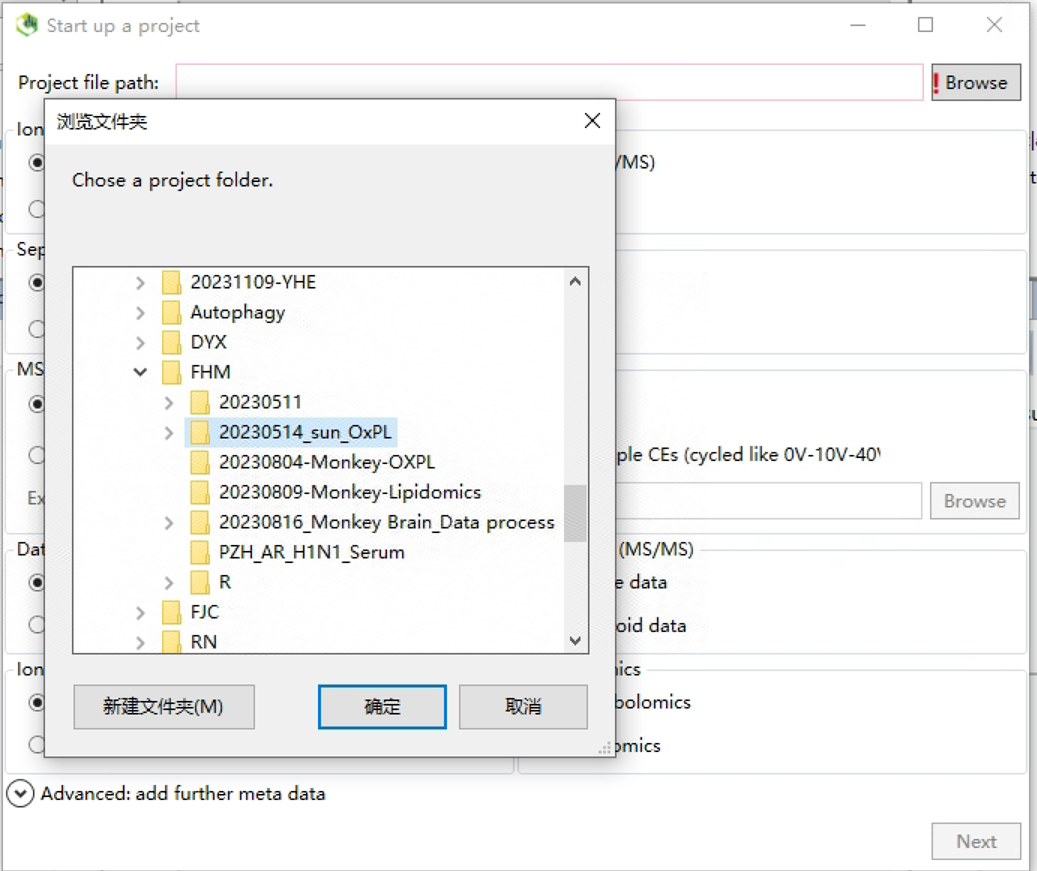
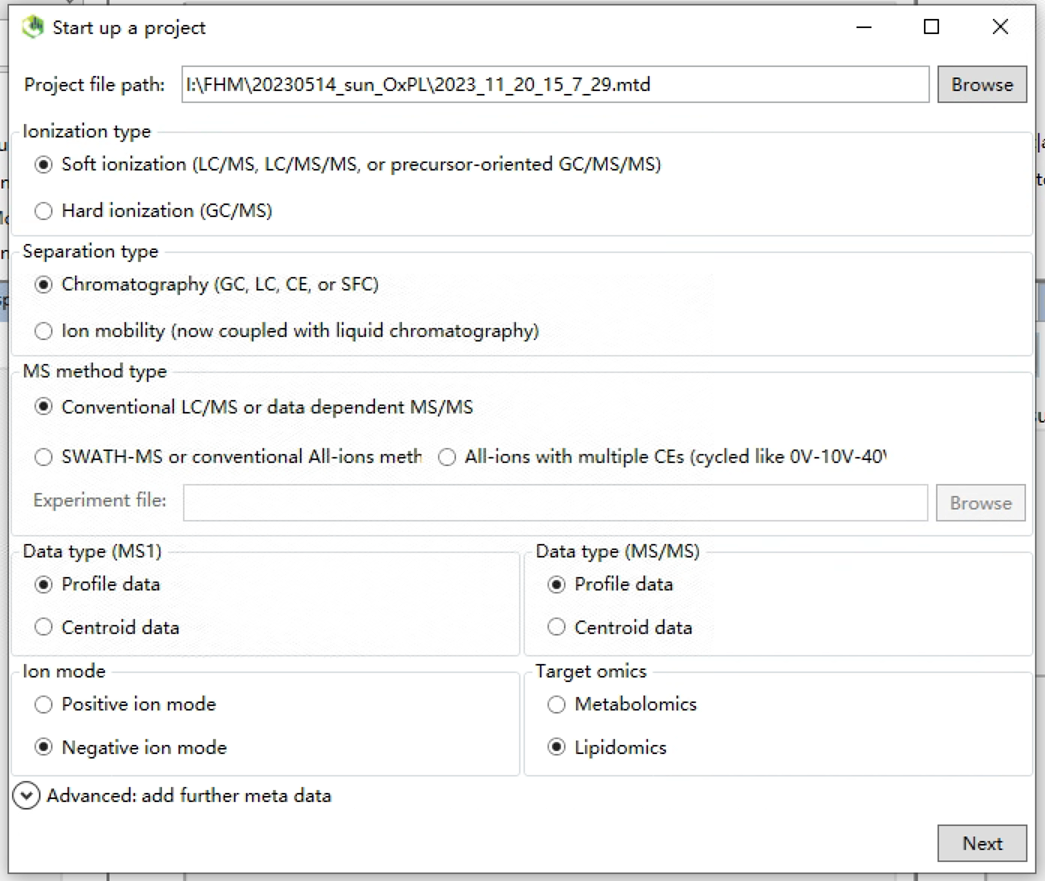
1.3 设置数据类型参数
Ionization type:LC/MS
Separation type:LC
MS method type:Conventional LC/MS or data dependent MS/MS
Data type:Profile
Ion Mode:POS/NEG
target omics:Metabolomics/Lipidomics
MSDIAL一次只能处理一种模式和一种组学。氧化磷脂组学只用处理负离子模式,脂质组学和代谢组学均需分别处理正负模式。设置好,点next。
1.4 导入数据
点Browse,弹出文件夹,显示空白,修改右下角的数据格式,选择.raw。导入序列中的监测QC、样品一级数据、QC二级数据。
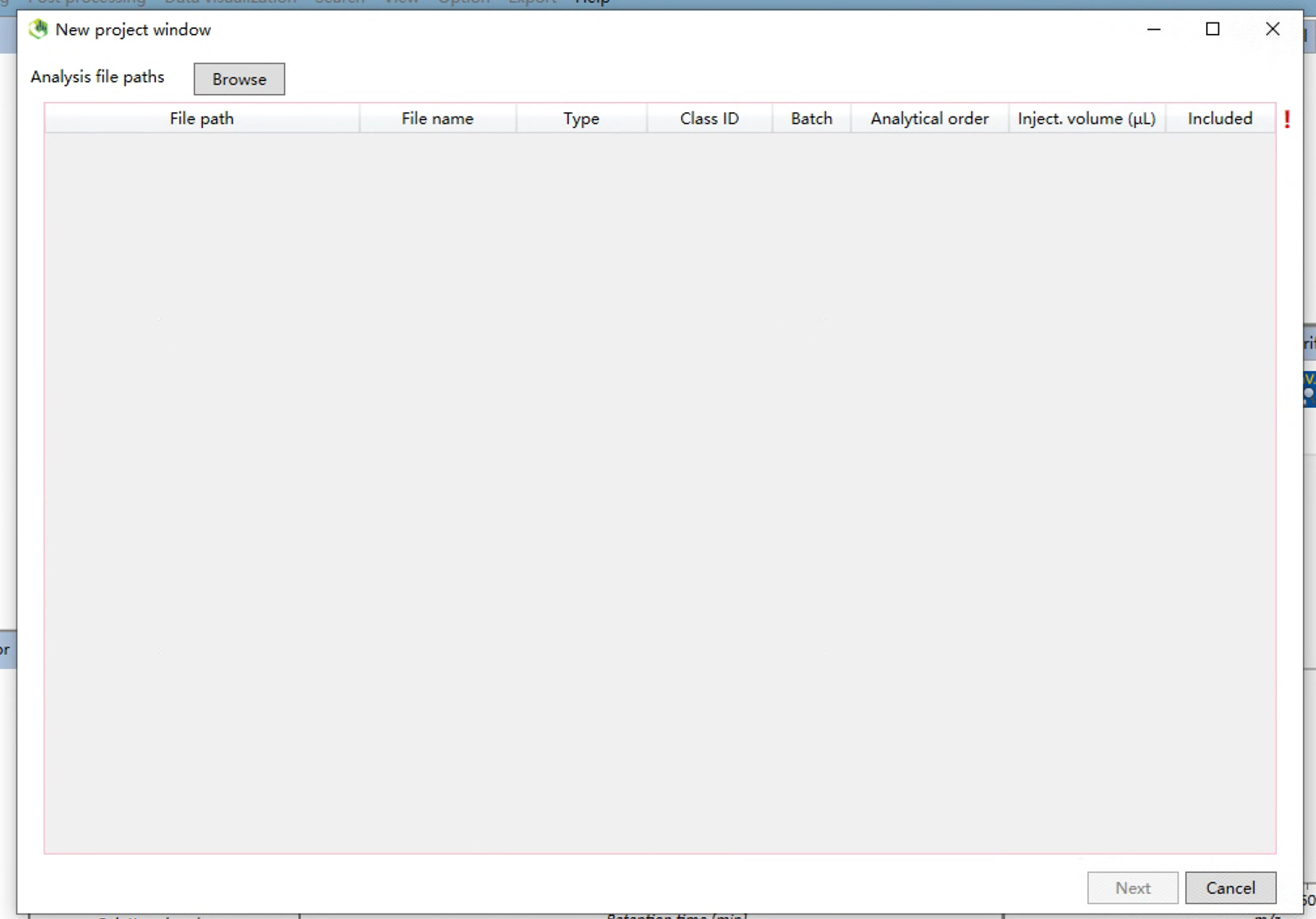
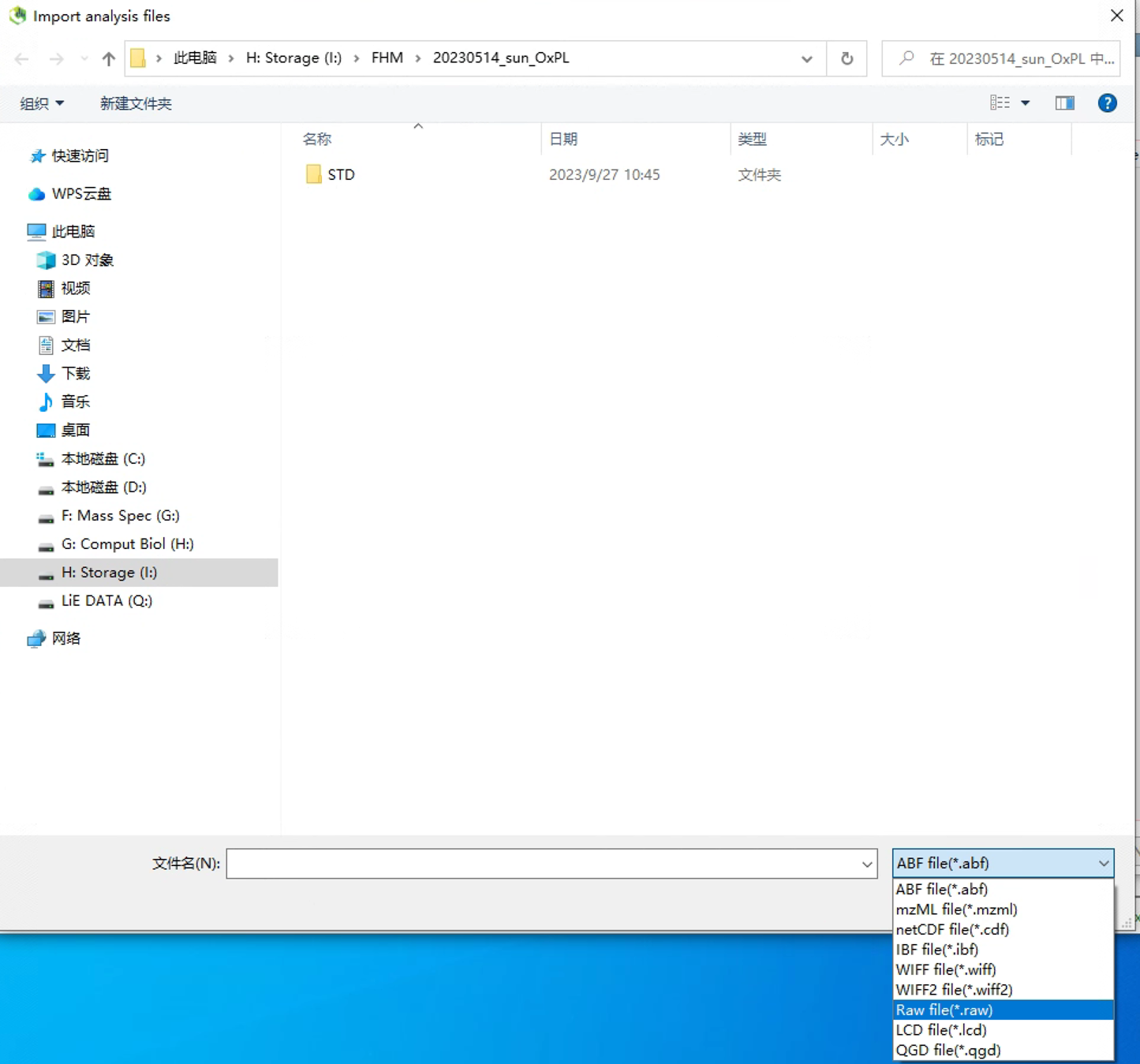
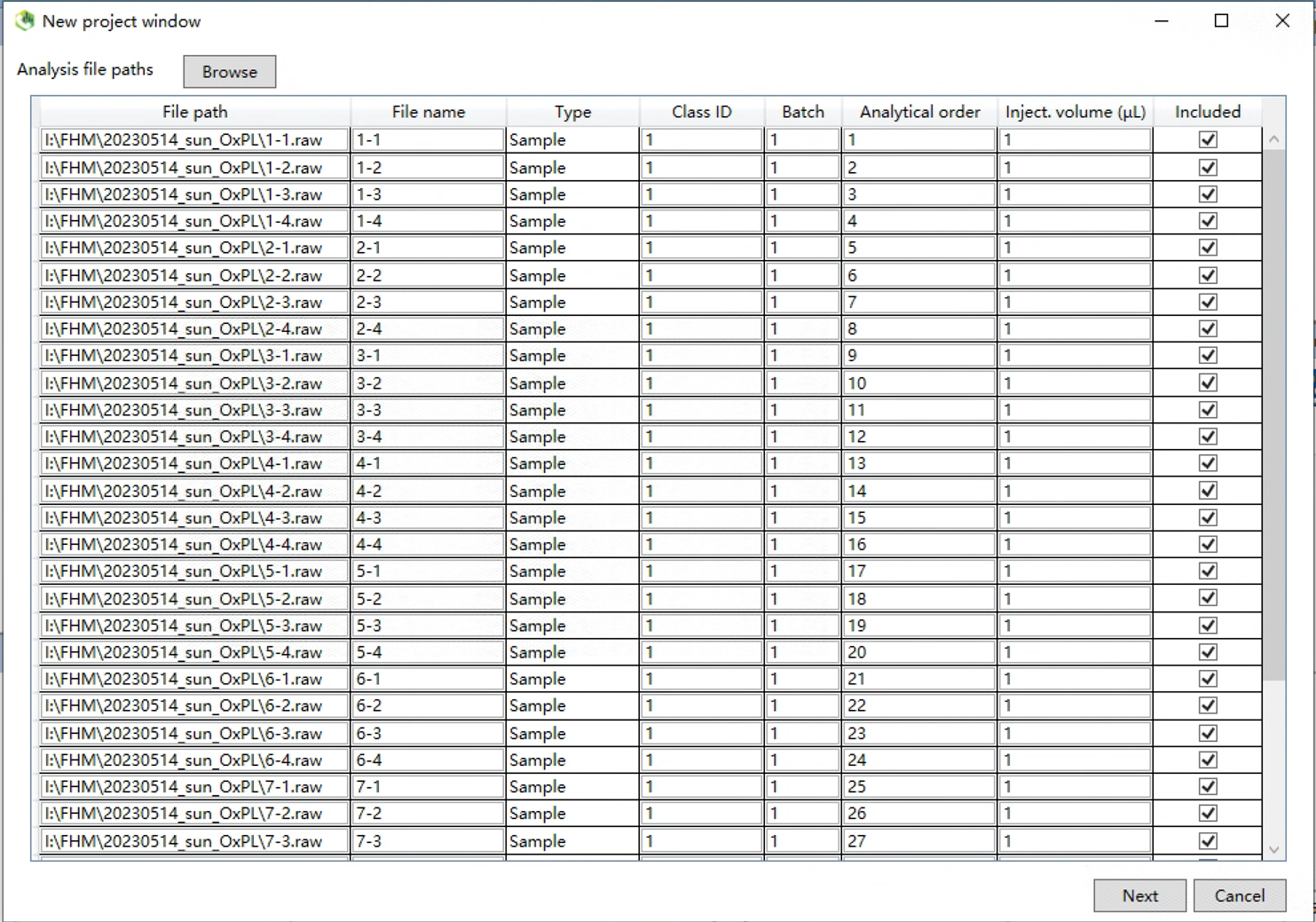
1.5 修改样品类型
把样品的Type改成Sample,QC的Type改成QC,其他不用设置,默认即可。
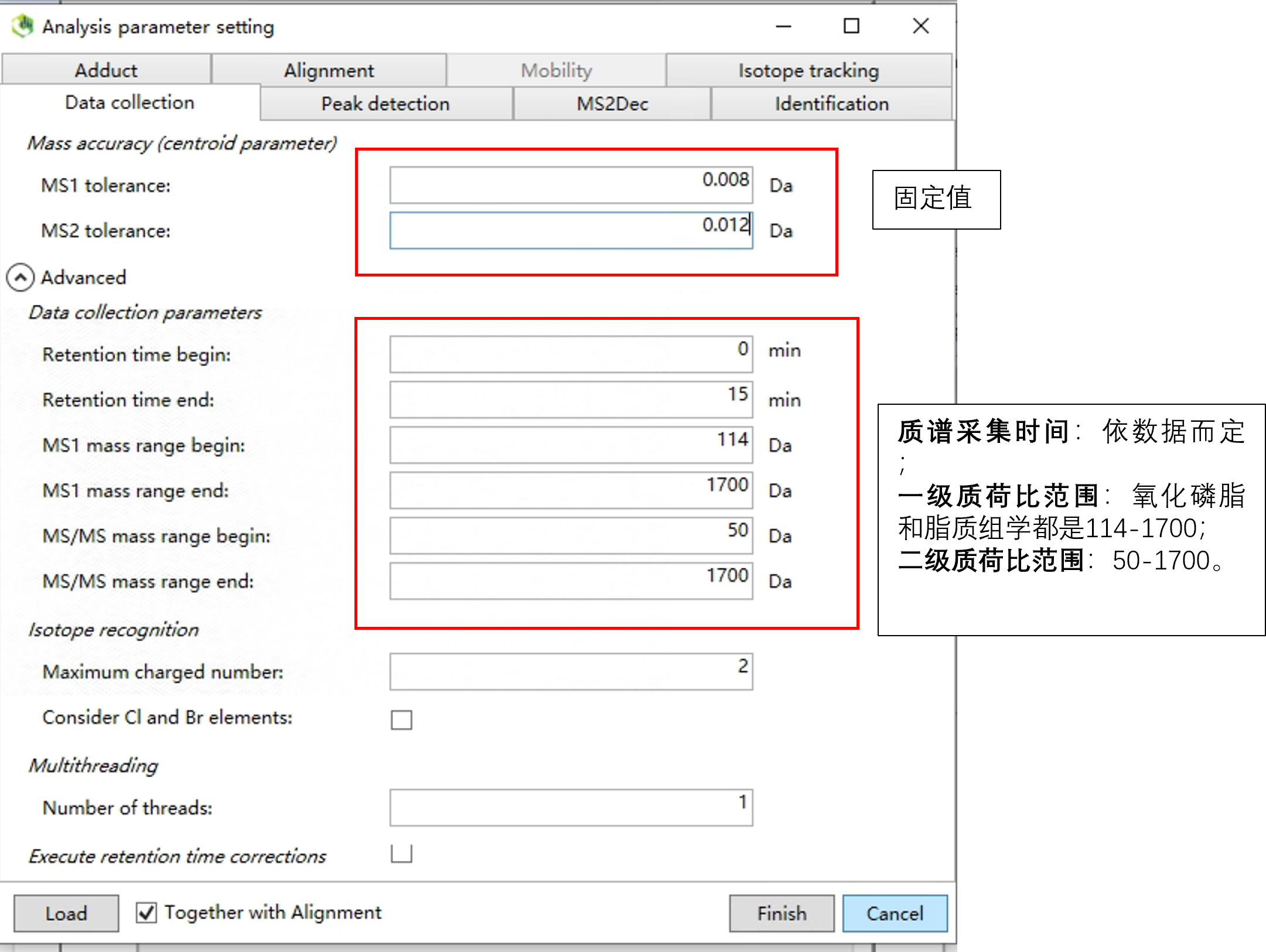
1.6 Data collection
除了上图中标出的地方,其他设置默认即可。
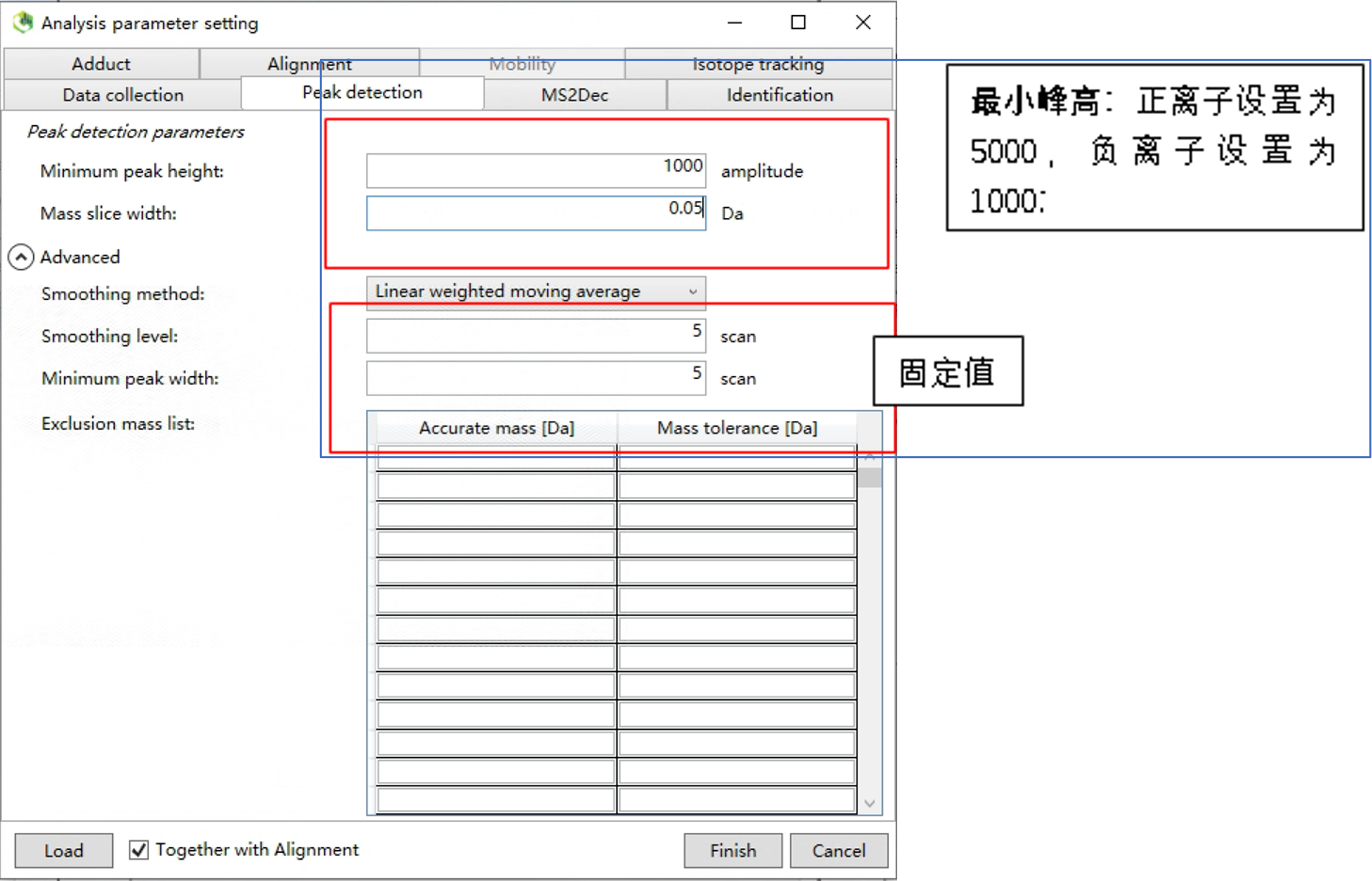
1.7 Peak detection
正负离子设置不一样。相当于信号的门槛,超过门槛,峰才会被监测到。
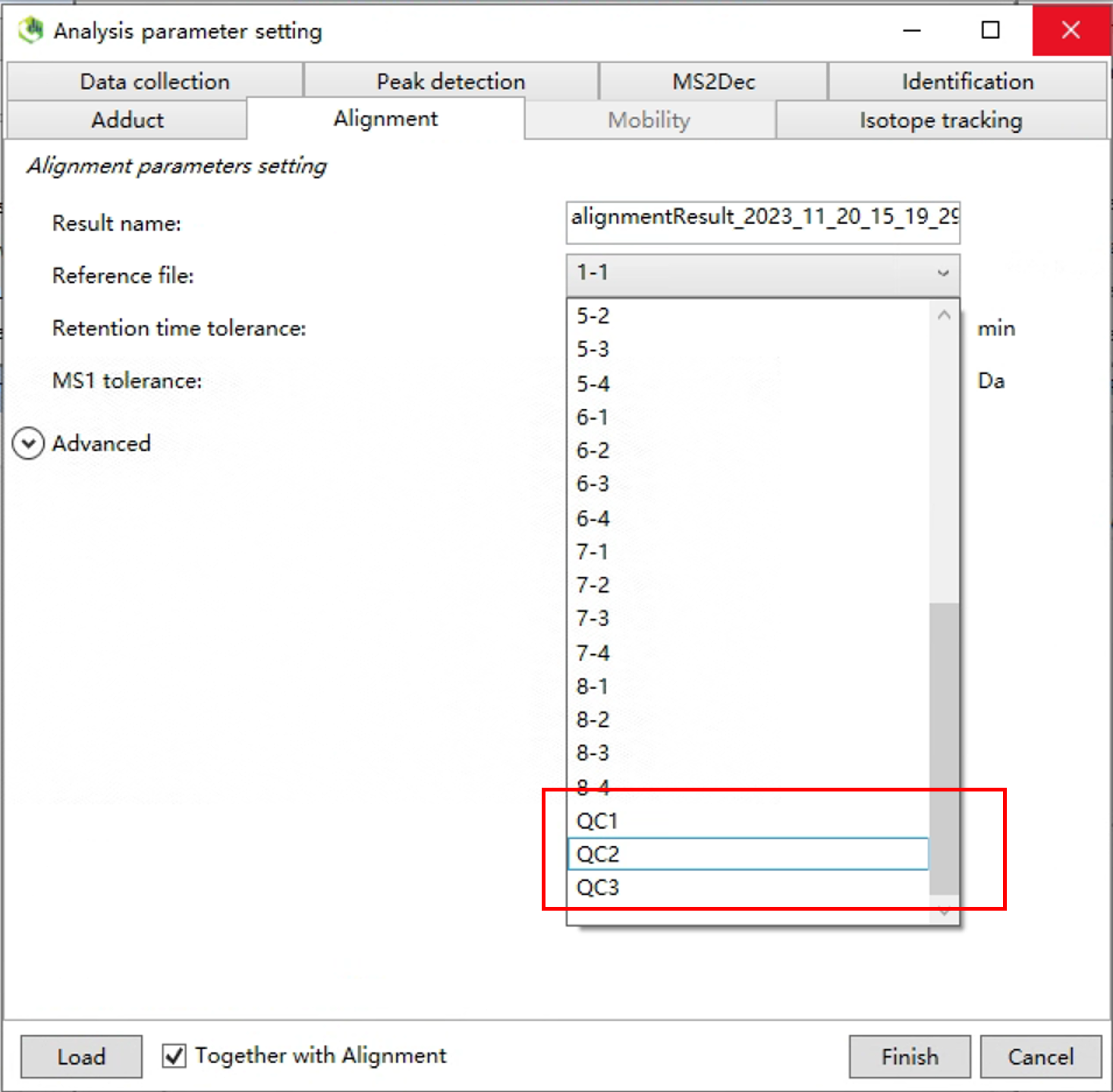
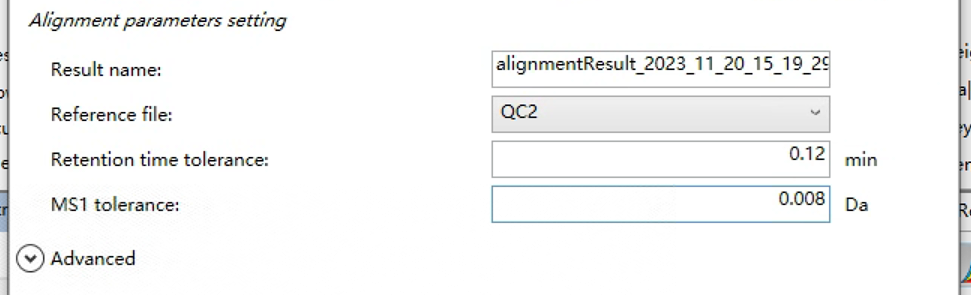
1.8 Alignment
对齐文件:选择几个监测QC里居中的监测QC,这是建立在数据稳定的条件下进行设置的。如果数据稳定,那么这三个QC应该是稳定的,浮动不大的,所以我们就可以选择居中的QC来让所有数据进行一个对齐。
RT的容差:一般来说设置0.12min即可(表示整体数据的RT偏移最大值和最小值之间不会超过0.24),如果想要处理结果更精确,就按照数据整体趋势来定,如果RT偏移最大值和最小值没有超过0.2,那么这里设置0.1min。按照数据的质量来调整参数,会让处理结果更好。
MS1质量容差:0.08(固定)
其他设置默认
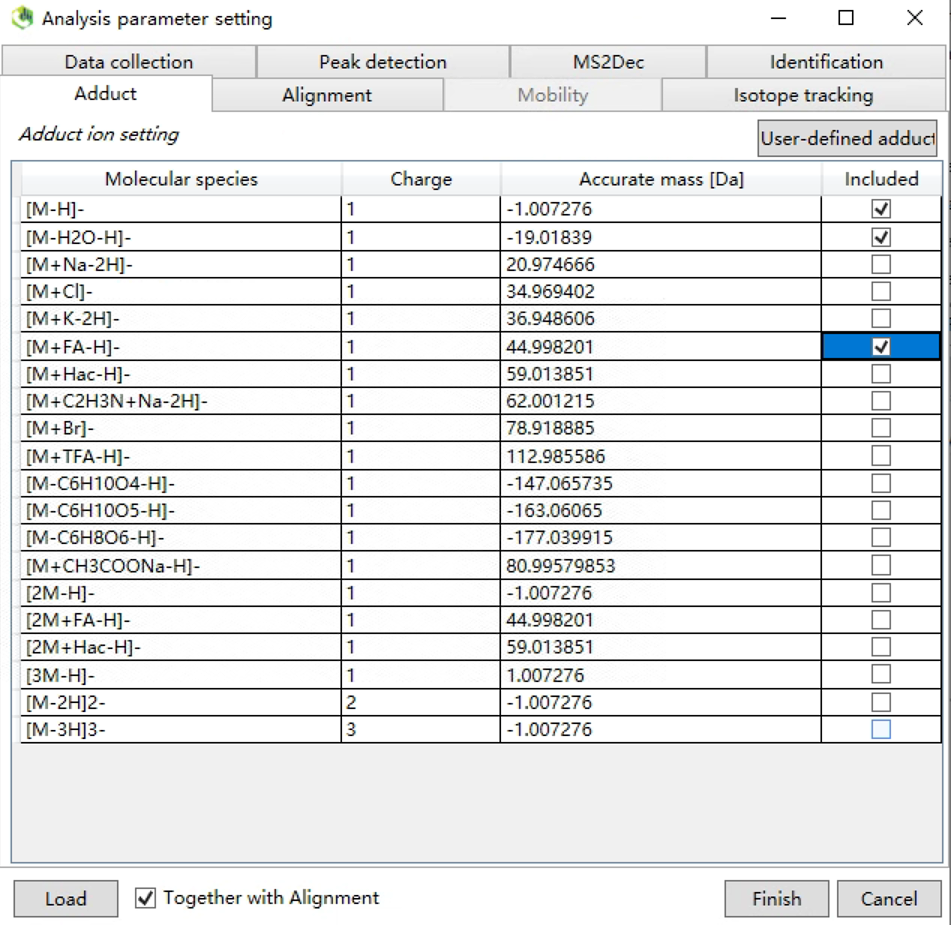
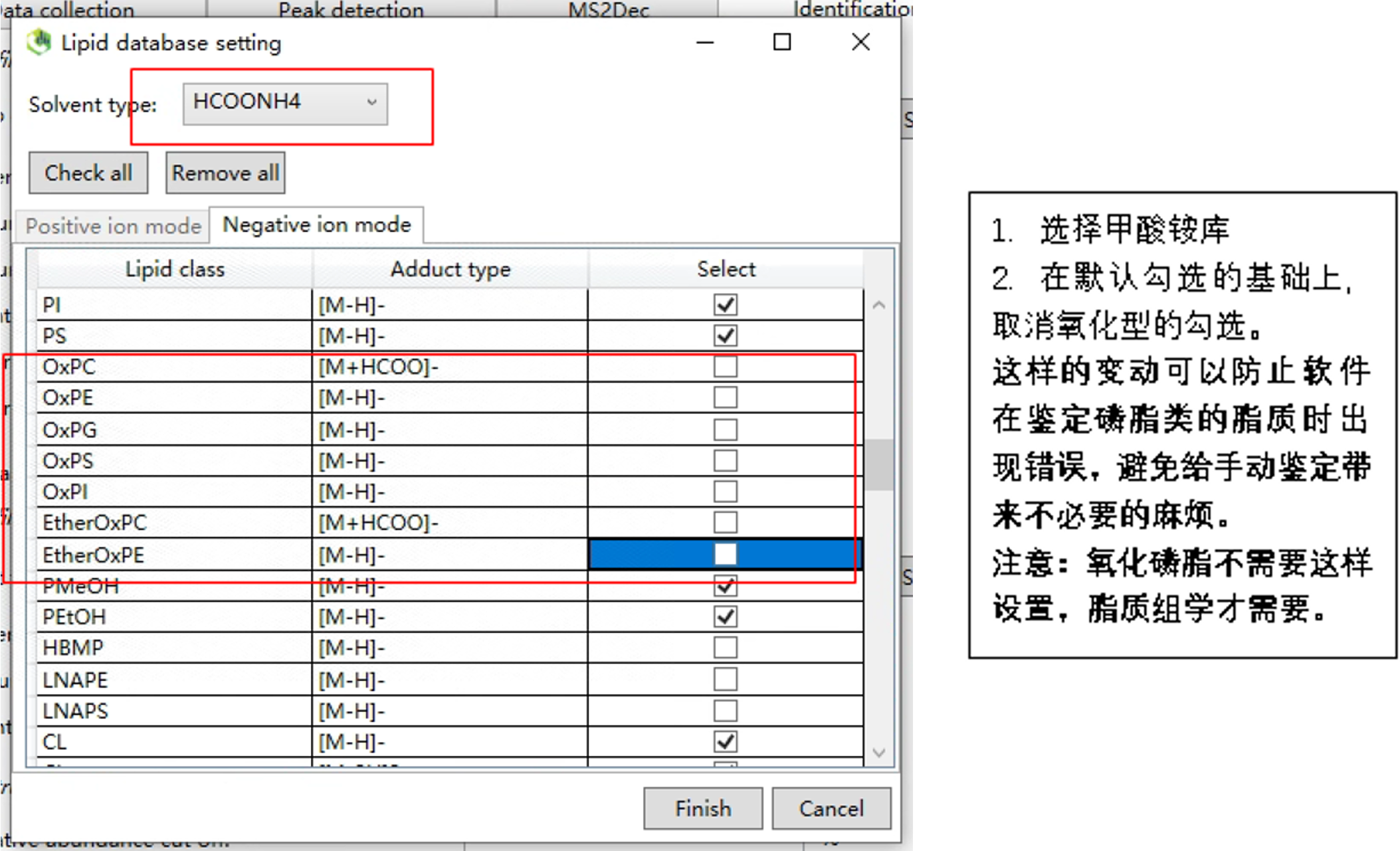
1.9 加合形式设置
依据流动相来定。一般来说氧化磷脂组学和脂质组学选择M-H、M-H2O-H、M+FA-H。
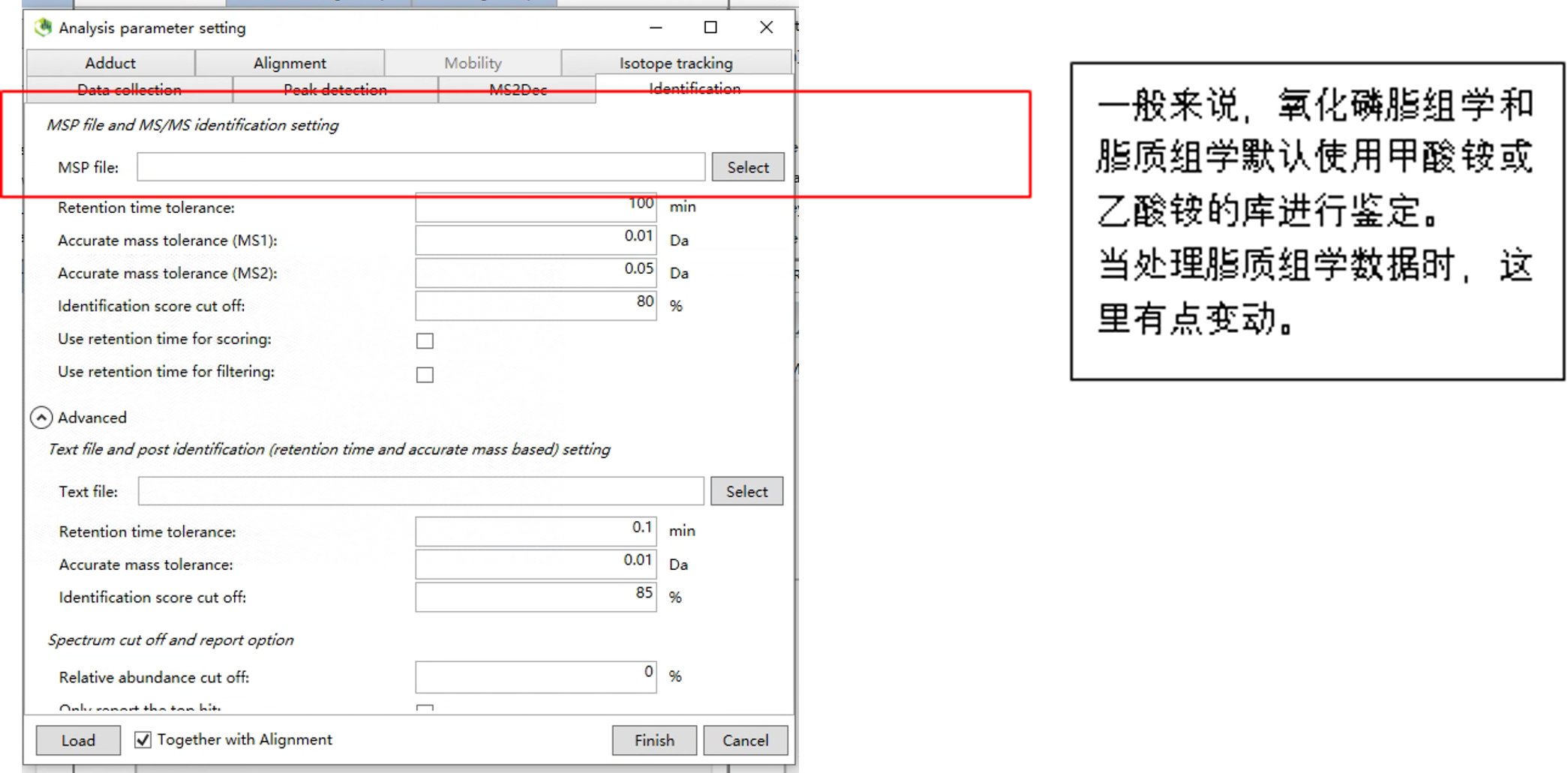
1.10 Identification
当处理代谢组学和氧化磷脂组学时,这个模块的设置不用变动,默认即可;当处理脂质组学时可以进行参考。
全部设置好以后就点finish,软件就开始run。
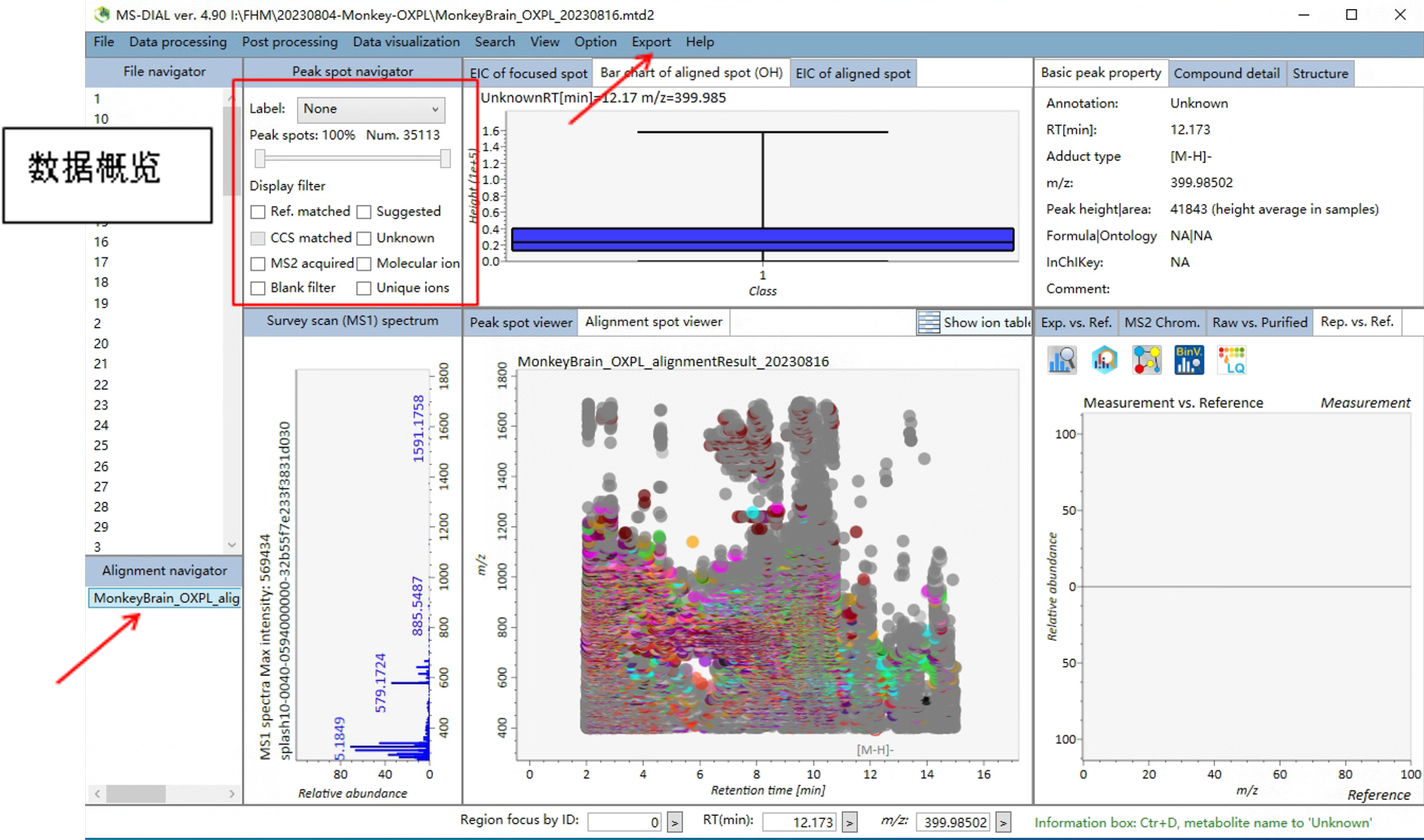
1.11 处理结束-导出表格
双击Alignment文件,就可以看到数据对齐后的样子。
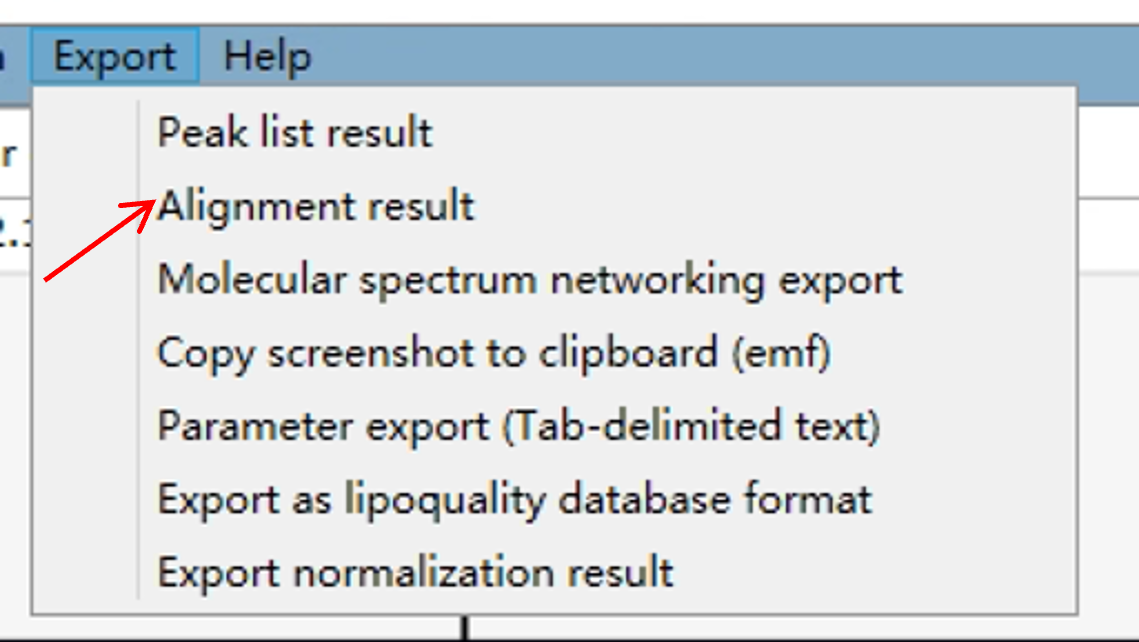
点击Export,点击Alignment result
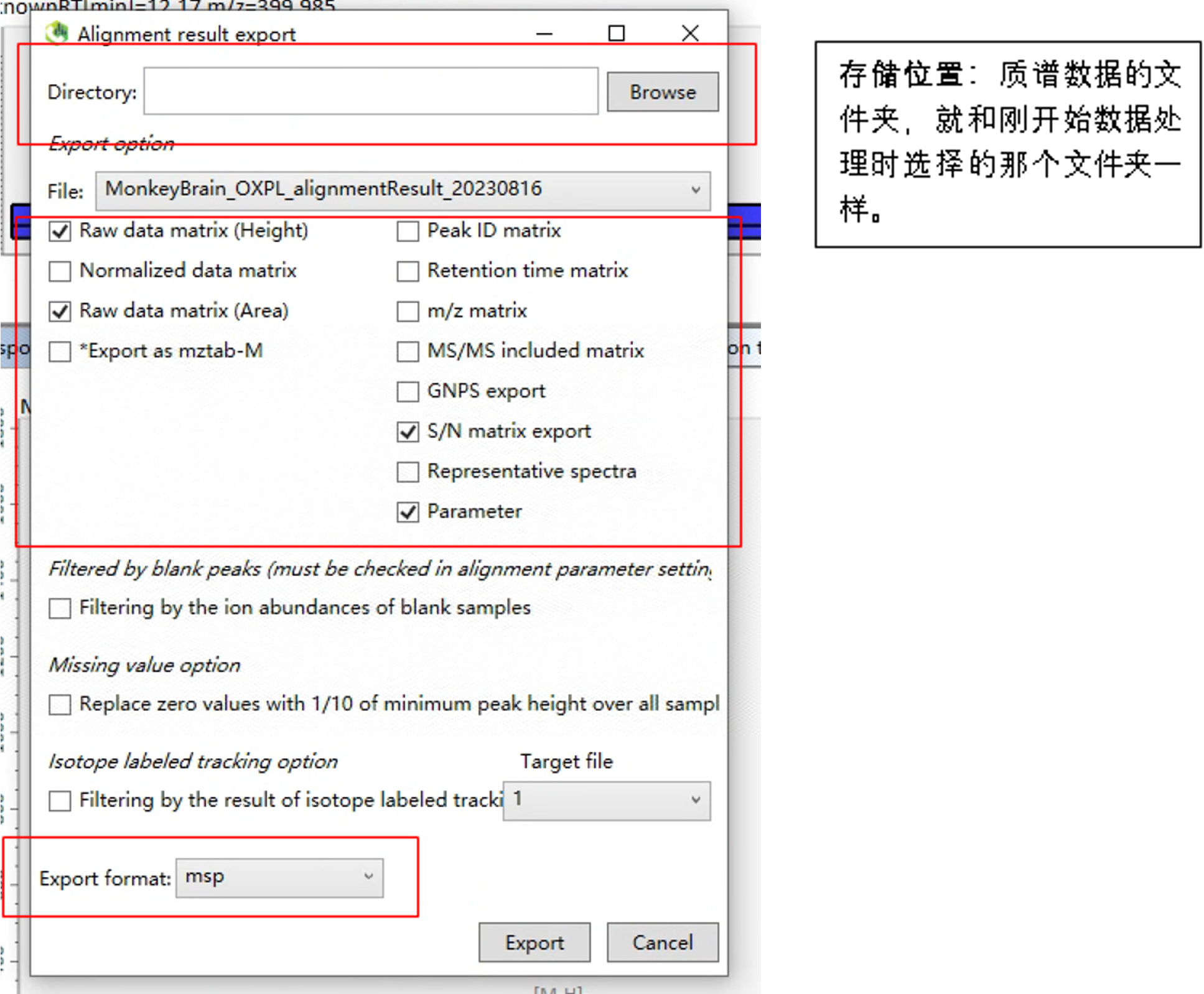
如果是氧化磷脂组学,我们需要进一步处理Area表格;如果是脂质组学,就可以把Area表格拿去做去重和手动鉴定了
2 使用Jupyter对Excel表格进行鉴定
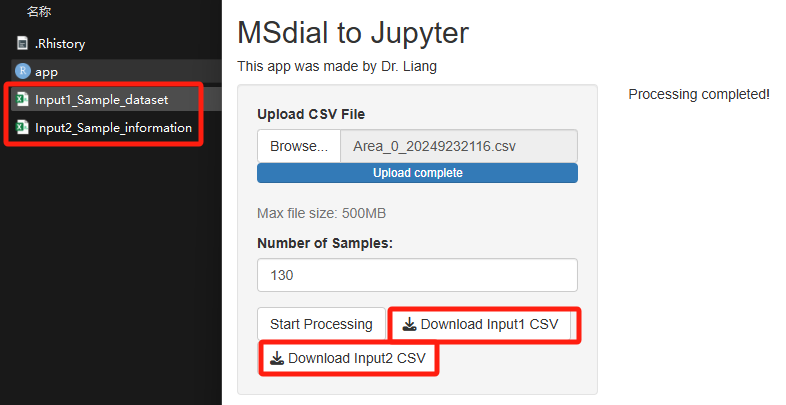
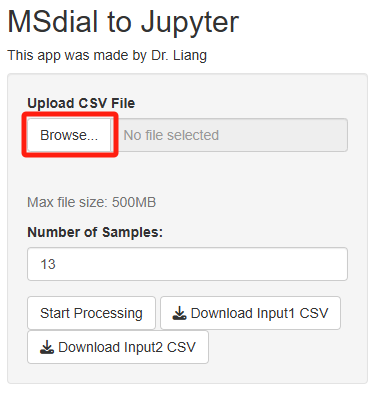
2.1 点击Browse加载MSdial导出的csv文件(通过txt转化)
2.2 上传成功后,更改样品数量为130个,点击Start Processing
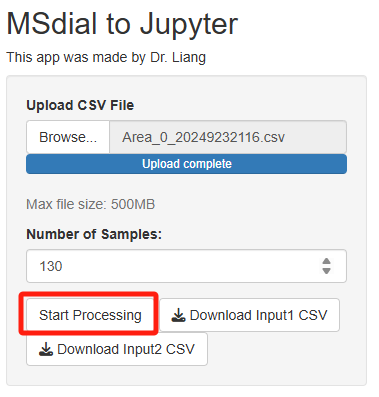
2.3 Processing completed完成,下载input1和input2